2004年由瑞士苏黎世联邦理工学院Dario Neri实验室提出(见图)。
首页
[{"ID":42422,"Name":"理学"},{"ID":62083,"Name":"化学"},{"ID":105196,"Name":"化学生物学"},{"ID":105479,"Name":"计算化学生物学"},{"ID":105480,"Name":"生物组学"},{"ID":105484,"Name":"[数据文库]"},{"ID":105488,"Name":"编码自组装分子库"}]
. 理学 . 化学 . 化学生物学 . 计算化学生物学 . 生物组学 . [数据文库] . 编码自组装分子库编码自组装分子库
/encoded self-assembling chemical(ESAC) library/
最后更新 2024-06-01
浏览 121次
一类基于碎片的DNA编码分子库。
- 英文名称
- encoded self-assembling chemical(ESAC) library
- 创建时间
- 2004
- 所属学科
- 化学
与其他DNA编码分子库由单一分子库构建不同,该类分子库由两个或两个以上亚分子库构建。每一个亚分子库由若干个连接结构碎片的编码DNA组成,其中碎片与编码DNA一一对应。在同一亚分子库中,编码DNA由聚合酶链式反应(PCR)引物结合区、编码区和互补配对区组成。PCR引物结合区与引物结合,实现筛选结果的扩增;编码区记录对应碎片的结构信息;互补配对区控制亚分子库中的编码DNA,使其通过互补配对原理与另一亚分子库中的编码DNA杂交,自组装后构成最终的分子库。如果分子库由两个亚分子库构成,其中一个亚分子库包含n个连接不同碎片的编码DNA;另一个亚分子库包含m个连接不同碎片的编码DNA,则通过DNA杂交后构建的编码自组装分子库包含n×m个碎片组合对。
使用靶标(通常是蛋白质)对自组装分子库进行基于亲和力的筛选,并通过PCR扩增与DNA高通量测序获得参与自组装组合的碎片结构信息。
构成潜在活性的碎片组合对,与不同的连接基团相连,构成碎片连接物。将这些连接物通过不同类型的生化手段,以用于活性测定。





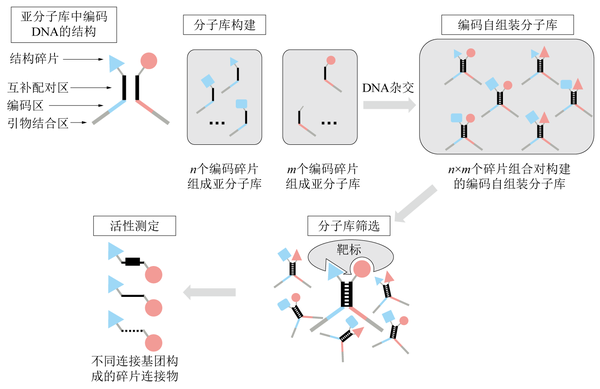 编码自组装分子库作用示意图
编码自组装分子库作用示意图
 京公网安备 11010202008139号
京公网安备 11010202008139号
